Der Einsatz der künstlichen Intelligenz schreitet auch in der Medizin immer stärker voran. Wir wollen heute ein konkretes Projekt vorstellen, wie mihilfe von KI Röntgenbilder ausgewertet werden können.
Diagnose zu Röntgenbilder mithilfe künstlicher Intelligenz
Heute wollen wir in Tun gehen und zeigen, wie jeder mit relativ einfachen Mitteln einen Prototyp zur Auswertung und Diagnose von Röntgenbildern mithilfe künstlicher Intelligenz erstellen kann.
Wir setzen dafür die fastai Libraray ein. Fastai ist ein aktuelles Deep Learning Framework basierend auf der Programmiersprache Python, das immer stärkere Aufmerksamkeit auf sich zieht.
Mit fastai können sehr einfach deep learning Modelle für einen großen Anwendungsbereich erstellt werden. Die Möglichkeiten erstrecken sich von tabellarischen Daten über textuelle Daten und Bilddaten. Seit der Version 1.0 gibt es mit dem Modul fastai.medical.imaging auch eine explizite Unterstützung für den Einsatz von medizinischen Bildern und künstlicher Intelligenz.
Im weiteren Verlauf dieses Blogartikels setzen wir diese Bibliothek von fastai ein, um einen Klassifizierer auf Basis eines deep learning Modells zu entwickeln, der Röntgenbilder in eine von zwei Klassen einteilt:
- pneumothorax
- no pneumothorax
Für alle, die sich über diese Einführung hinaus in das fastai Framework einarbeiten wollen, sei unser online Kurs zu künstlicher Intelligenz “welcome2ki Teil 3 - Deep Learning Projekte selbst umsetzen”. In diesem online Training gehen wir auf alle erforderlichen Themen ein, um selbst mithilfe von deep learning und künstlicher Intelligenz KI-Systeme zu programmieren. Wir besprechen die Erstellung und Bereinigung von Trainingsdaten, die Entwicklung eine KI-Modells und das Deployment für den Enduser.
Die Trainingsdaten - DICOM Bilddaten
Für unser KI-Modell zur Anwendung in der Medizin verwenden wir ein Datenset, das gemeinsam mit dem fastai Framework ausgeliefert wird: SIIM Small. Dieses Datenset enthält 250 DICOM Bilder mit Röntgenaufnahmen der Lunge.
Bevor wir daher auf die Erstellung unseres deep learning Modells näher eingehen, wollen wir an dieser Stelle den DICOM Standard für medizinische Bilddaten näher beschreiben.
Der DICOM Standard für medizinische Bilddaten
Das Akronym DICOM steht für digital imageing and comunnication in medicine. DICOM ist der defacto Standard für die Speicherung und Übertragung von Bilddaten in der Medizin, wie Röntgenaufnahmen, CT oder MRT-Bilder oder Ultraschall.
Aus diesem Grund ist es wichtig, zumindet über ein Basiswissen im Umgang mit DICOM Bildern zu verfügen, wenn man künstliche Intelligenz im Bereich der bildgebenden Medizin einsetzen möchte.
Dadurch, dass DICOM von praktisch allen Geräteherstellern als Standardformat akzeptiert wurde, ermöglicht der Standard die geräteunabhängige Übertragung von Bildern zwischen Geräten unterschiedlicher Hersteller. DICOM Bilder werden typischerweise in Bilddatenbanken mit der Bezeichnung PACS - picture archiving and communication system gespeichert.
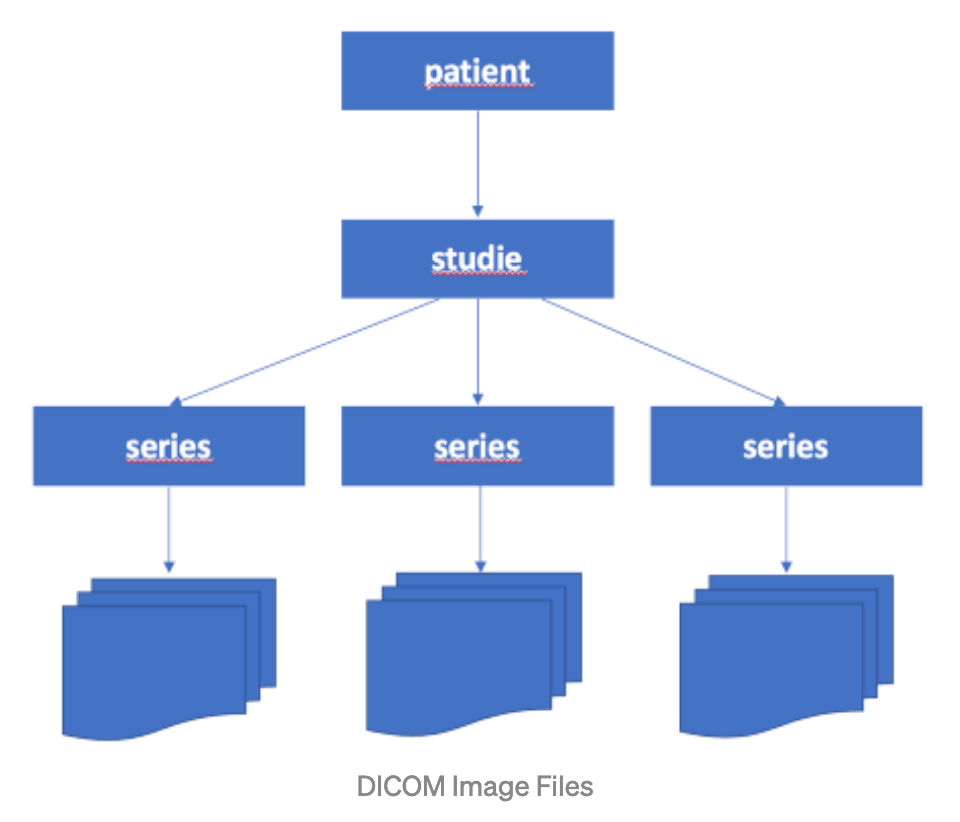
Die Abbildung zeigt die hierarchische Struktur von DICOM Dateien. Zu einem Patienten wird eine Studie zugewiesen. Das kann etwa ein bestimmter Besuch in der radiologischen Ordination sein. Unterhalb dieser Studie können beliebig viele Serien stehen. Diese sind die einzelnen Untersuchungen. Pro Untersuchung kann eine ganze Bildserie angefertigt werden.
Zusätzlich zu den eigentlichen Bilddaten können in DICOM Dateien auch weitere Attribute gespeichert werden - Metainformationen über das jeweilige Bild.
Diese Attribute werden wiederum in Module unterteilt. Module können einzelne Daten enthalten oder auch gruppierte Objekte, die dann auch wieder an anderen Stellen der Attribute wiederverwendet werden können.

DICOM Bilder weisen für die einzelnen Pixelwerte typischerweise einen Wertebereich zwischen -32768 und +32768 auf, während klassische 8 Bit Graustufenbilder nur einen Wertebereich zwischen 0 und 255 aufweisen.
Die Pixelwerte im DICOM Standard entsprechen den Werten auf der Hounsfield Skala, welche die Materialdichte angeben. Wasser hat typischerweise Werte rund um 0 HE (Hounsfield Einheiten), Luft -1000 HE.
Durch Gruppierung der Pixelwerte in bestimmte Wertefenster können die Bilder für das menschliche Auge besser aufbereitet werden, da dieses nur etwa 17 unterschiedliche Graustufen unterscheiden kann.
Das Fastai SIIM Small DICOM Trainingsdatenset
Das im fastai Framework mitgelieferte DICOM Datenset beinhaltet 250 Röntgenaufnahmen inkl. der dazugehörigen Lables (groundtruth). Dieses DICOM Datenset wird direkt über die URLs in der Fastai Library angesprochen.
Es beinhaltet jedoch kein RescaleIntercept und RescaleSlope, mit denen die Pixelwerte auf die Hounsfield Skala umgerechnet werden können.
Die Bereiche für die Pixel sind mit einem Wertebereich von 0 bis 255 beschränkt und entsprechen damit einfachen Graustufenbildern.
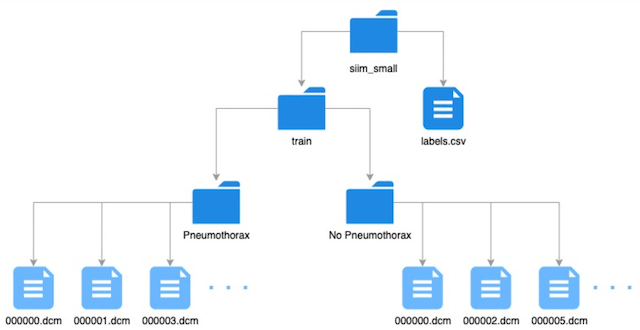
Wir finden die Trainingsdaten also in zwei verschiedenen Verzeichnissen, die nach den entsprechenden Labels benannt sind. Zusätzlich finden wir eine csv Datei, die ebenfalls die Werte für die abhängige Variable unseres deep leanring Modells beinhaltet.
Laden der Daten:
pneumothorax_source = untar_data(URLs.SIIM_SMALL)
Laden der Labels für unser deep learning Modell.
pneumothorax_source = untar_data(URLs.SIIM_SMALL)
Aufbau der Daten für unser KI-Modell
Wir verwenden die DataBlock API aus dem fastai deep learning Framework und erstellen einen individuellen DataBlock, der unsere DICOM Bilder klassifiziert.
pneumothorax = DataBlock(blocks=(ImageBlock(cls=PILDicom), CategoryBlock),
get_x=lambda x:pneumothorax_source/f"{x[0]}",
get_y=lambda x:x[1],
batch_tfms=[*aug_transforms(size=224),
Normalize.from_stats(*imagenet_stats)])
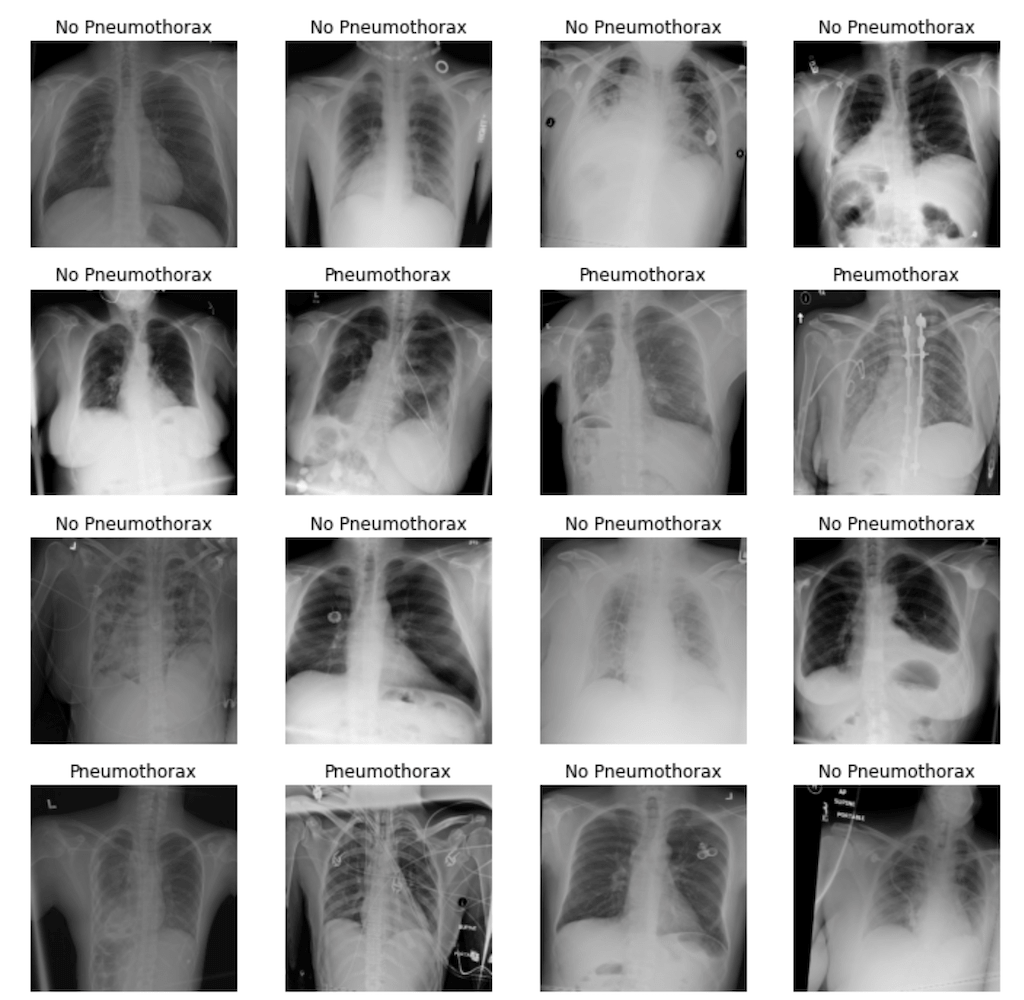
Auf Basis dieses DataBlocks können wir nun unsere Dataloader erstellen. Wir lassen uns einen zufälligen Batch aus unseren Trainingsdaten anzeigen, um diese vor dem Einsatz mit unserem KI-Modell noch einmal überprüfen zu können.
dls = pneumothorax.dataloaders(df.values, num_workers=0)
dls = pneumothorax.dataloaders(df.values)
dls.show_batch(max_n=16)
Trainieren unserer künstlichen Intelligenz für die Klassifizierung von Röntgenaufnahmen
Wir erstellen als erstes unser Learner-Objekt, das die gesamte Modellarchitektur enthält.
learn = cnn_learner(dls, resnet34, metrics=accuracy)
Da wir keine eigenen Werte für die Loss-Funktion und die Optimizer-Funktion angegeben haben, versucht fastai, diese aus der Struktur unserer Trainingsdaten abzuleiten. Wir können jedoch jederzeit anhand des jeweiligen Properties überprüfen, welche Wahl das fastai deep learning Framework getroffen hat.
learn.loss_func
FlattenedLoss of CrossEntropyLoss()
learn.opt_func
<function fastai.optimizer.Adam>
Nun können wir unser Modell bereits trainieren….
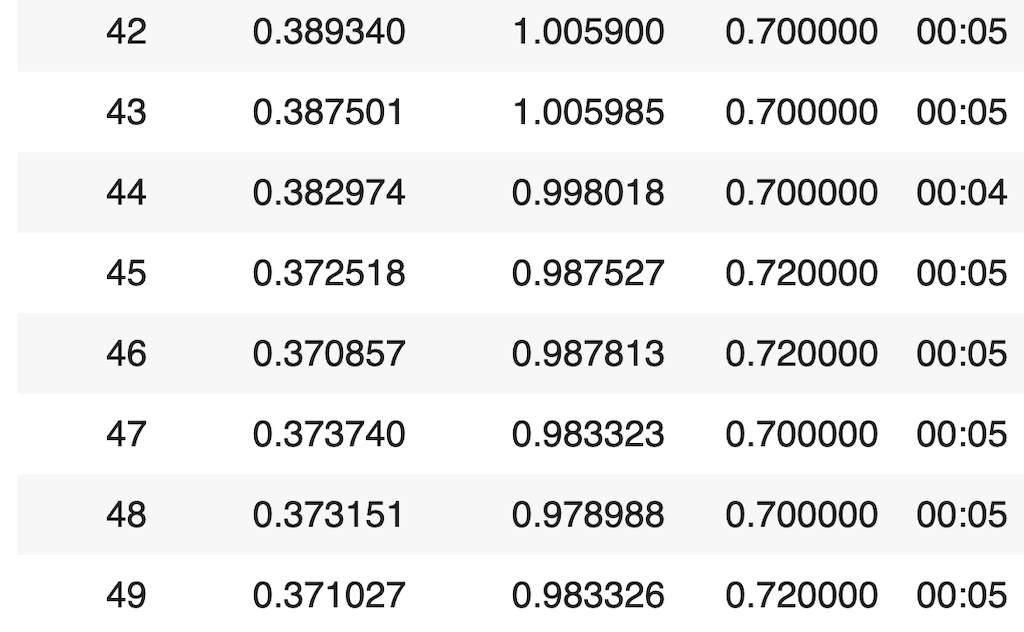
learn.fit_one_cycle(50)
Wir sehen, dass unser KI-Modell in den letzten epochs bereits eine Genauigkeit (Accuracy) von über 70% erreicht hat, was für einen ersten Prototypen nicht ganz schlecht ist.
Das einfachste, anzunehmende Referenzmodell, wäre einfach eine zufällige Klasse auszuwählen, was langfristig zu einer Genauigkeit von ca. 50% führen wird (vorausgesetzt, dass beide Klassen etwa gleich häufig vorkommen.)
Prognosen unseres Deep Learning Modells
Nun wollen wir uns einige Prognosen unseres Deep Learning Modells anhand der Testdaten ansehen, um entscheiden zu können, wie gut unsere künstliche Intelligenz für Radiologie tatsächlich funktioniert.
learn.show_results(max_n=16)
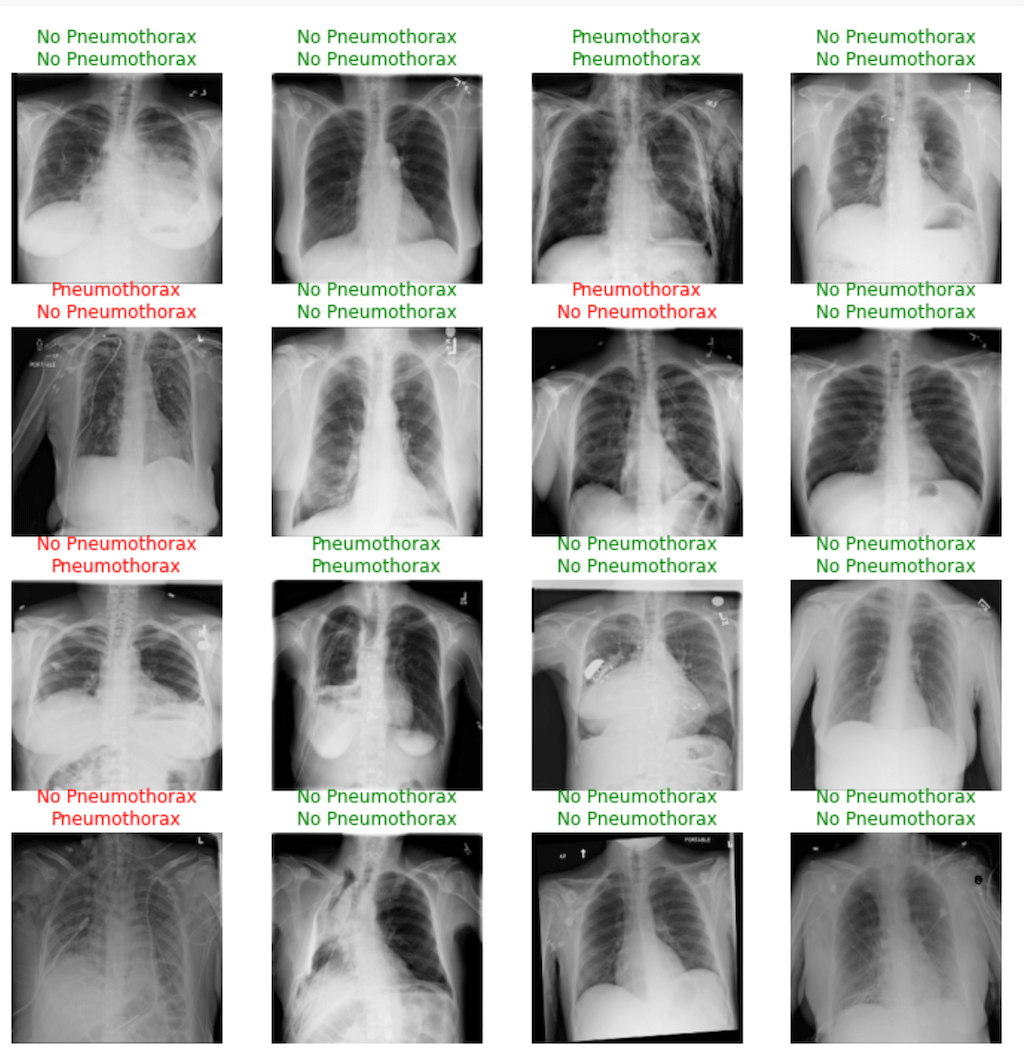
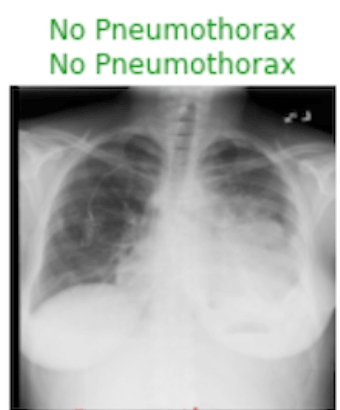
Hier sehen wir immer in grün die beiden Labels (Groundtruth und Prognose unserer künstlichen Intelligenz), wenn unser deep learning Modell richtig lag und die beiden Labels in Rot, für jene Fälle, bei denen unser Modell daneben lag.
Fazit - Künstliche Intelligenz in der Radiologie
Wir haben mit sehr wenigen Trainingsdaten und wenig Programmieraufwand ein Deep Learning Modell für die Medizin entwickelt, das bei zwei Klassen eine Genauigkeit von über 70% erreicht. Das ist kein schlechter Ausgangspunkt.
Für den tatsächlichen Einsatz als Diagnosewerkzeug in der Medizin müssen wir jedoch noch mehr Aspekte berücksichtigen. Hier geht es um Erkrankungen von Menschen, bei denen wir weder falsch-positive, noch falsch-negative Ergebnisse erzielen wollen. Aus diesem Grund kann (unser) Modell den Radiologen sicher nicht ersetzen. Als Assistenzsystem für den Radiologen oder Triage-System, welche Bilder am schnellsten durch den Arzt diagnostiziert werden sollten, sind derartige Systeme jedoch am breiten Markt im Einsatz.
Allen die tiefer in die Materie der künstlichen Intelligenz einsteigen wollen und selbst aktiv werden wollen, empfehlen wir unsere Akademie für künstliche Intelligenz (nicht nur für die Medizin) unter https://academy.we-make.ai.
Literaturempfehlung DICOM: Der DICOM Standard in der Kardiologie als Basis für die Interoperabilität medizinischer Bildsysteme und die Entwicklung eines vernetzten Bildarchivs
